登陆查看更多优秀资源帖,与同道便捷交流讨论
您需要 登录 才可以下载或查看,没有帐号?立即注册
x
Alphafold 3,一种新的革命性的人工智能 (AI) 模型,将以前所未有的准确性预测包含更广泛的生物分子,包括配体、离子、核酸和修饰残基的复合物的结构。与现有预测方法相比,Alphafold3 预测蛋白与其他生物分子相互作用的准确性有至少 50% 的提升,对于某些重要的相互作用类别,预测准确度甚至翻了一番。Alphafold3 架构Alphafold3 是通过对 AlphaFold 2 架构和训练过程的重大演进实现的,既适应更一般的化学结构,又提高了学习数据的效率。该系统通过用更简单的 Pairformer 模块替换 AlphaFold 2 的 Evoformer 来减少多序列比对(MSA)处理的数量。此外,它通过使用扩散模块直接预测原子坐标,取代了 AlphaFold 2 中作用于氨基酸特定框架和侧链扭转角的结构模块。扩散过程的多尺度性质(低噪声水平促使网络改善局部结构)还使我们能够消除立体化学损失,并且在网络中大部分特殊处理键合模式,轻松适应任意化学组分。 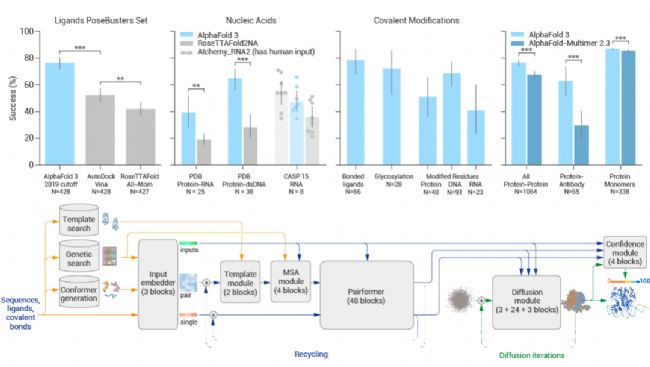 图 2. Alphafold3 可以准确预测复合物的结构以及 Alphafold3 架构[1]
Alphafold3 可以准确预测复合物结构AlphaFold3 可以将蛋白质与其相应的配体作为输入一起折叠,并理解它们的作用机制。让我们一起通过如下三个示例,来看看 AlphaFold3 在哪些研发领域有新的突破。 首先,AlphaFold3 在预测蛋白-蛋白相互作用方面有很大改进。如图 2 展现了AlphaFold3 可以准确地预测 AziU3/U2 蛋白如何与底物结合,AziU3/U2 结构结合后会产生一个新的蛋白折叠。基于该案例,AlphaFold3 预计可以帮助我们理解更多蛋白-蛋白相互作用机制,将为开发小分子 PPI 抑制剂打开了新的局面。
 图 2. AlphaFold3 准确预测 AziU3/U2 蛋白与底物结合[2] 灰白色为实验测定复合物结构,淡蓝及淡粉色为预测结构 AlphaFold3 预测别构口袋的能力也同样值得期待。图 3 展现了 AlphaFold3 可以准确预测 PI5P4K 激酶的别构口袋以及和小分子的相互作用模式。别构类药物具有许多的优势,比如更好的特异性,较少的副作用以及克服耐药性等,相信 AlphaFold3 将为开发别构药物提供更多的见解。 图 3. AlphaFold3准确预测 PI5P4K 激酶的别构口袋以及和小分子的相互作用模式[2] 灰白色为实验测定复合物结构,淡蓝及淡粉色为预测结构 AlphaFold3 在膜蛋白领域的预测能力同样超群。如图 4,AlphaFold3 可以准确的预测 PORCN (膜蛋白)、LGK974 和 WNT3A 复合物的结构。一直以来,膜蛋白的结构转变在跨膜信号传递中发挥核心作用,但其结构复杂,难以预测,AlphaFold3 为膜蛋白的合理药物设计提供了可能。  图 4. AlphaFold3 准确预测 PORCN-LGK974-WNT3A 复合物-结构[2]灰白色为实验测定复合物结构,淡蓝及淡粉色为预测结构 如何使用 Alphafold3 预测结构图 5. AlphaFold Server 操作界面 该界面操作简单,第一步先从 PDB 或者 UniProt数据库找到相应的序列,然后逐一添加到相应的输入框,提交任务即可。小 M 测试了 PRMT5/MPE50 和 MTA 的复合物体系,如下动图展示,两个蛋白的相互作用界面以及 MTA 小分子的结合位点基本与解析的共晶结构(PBD ID:7S1Q)一致,预测结构和实验结构叠合后的 RMSD为 0.9,Alphafold3 实测好用。 相信 AlphaFold3 的出现,将为基于结构的药物筛选带来更大的机遇。MCE 拥有丰富的化合物库数据资源、高性能的计算机服务器,可提供专业的分子对接、虚拟筛选,分子动力学模拟服务,将竭诚为广大科研客户提供优质的早期药物研发服务!
共价化合物库
MCE 共价化合物库收录了 1600+ 种小分子抑制剂,包括已知共价抑制剂及具有共价反应基团的活性小分子化合物,如包含丙烯酰胺、活化末端乙炔、磺酰氟化物/酯、氯乙酰胺、烷基卤化物、环氧化物、氮杂环胺、二硫化物等,是筛选共价化合物,研究共价作用机制的有利工具。
| 共价化合物库 Plus
MCE 共价化合物库 Plus 收录了 2900+ 种小分子抑制剂,包括已知共价抑制剂及具有共价反应基团的活性小分子化合物,是筛选共价化合物,研究共价作用机制的有利工具。
MCE 共价化合物库 Plus 是对 MCE 共价化合物 (HY-L036) 的补充,增加了一些具有共价弹头的片段分子,具有更强的筛选能力。
| 半胱氨酸靶向共价化合物库
收录了4200+ 种包含不同靶向半氨酸共价弹头的小分子化合物。MCE半胱氨酸靶向共价化合物库主要使用以下共价弹头设计而成:Acrylamides、Propiolic acid ester、 Dimethylamine functionalized acrylamides 等。
| 半胱氨酸靶向共价片段库
收录了 3000+ 种包含不同靶向半氨酸共价弹头的片段化合物。MCE 半胱氨酸靶向共价片段库主要使用以下共价弹头设计而成:Acrylamides、Propiolic acid ester、 Dimethylamine functionalized acrylamides 等。所有片段均符合 RO3 原则,可以用于基于片段的共价药物设计。
|
|